上海交大毛亚飞团队与李卫东团队联合发文精准表征人类完整基因组中的大尺度结构变异
2023年7月4日,上海交通大学李卫东课题组硕士生王宣凯在Genome Biology发表题为Characterization of large-scale genomic differences in the first complete human genome的研究论文,比较分析了T2T-CHM13完整基因组与当前人类参考基因组模版(GRCh38)之间的大规模基因组差异,通过整合基因组学方法系统性地表征了两个人类基因组组装之间的大型结构变异(≥10 kbp),发现多个全新结构变异位点,并发现相关位点具有重要生物学功能,与脑疾病息息相关,该研究为后续的脑疾病精准医疗提供遗传基础[4]。
在发布第一份人类基因组草图20年后,Telomere-to-Telomere(T2T)联盟组装了世界上第一个完整基因组(T2T-CHM13)。T2T基因组完整表征了单个人类基因组所有序列,旨在提供对人类基因组更全面和准确的描述。GRCh38是现有的人类基因组模版,在多数科学研究中被广泛应用(如:关联分析,疾病风险位点分析,演化分析等)。然而,现有的人类基因组模版(GRCh38)在组装中存在大量的未知序列且多数复杂区域组装并不完整准确。因此,T2T基因组相对于GRCh38基因组提供了更完整、更准确的基因组序列,具有更高的连续性和精确性。未来随着T2T基因组的进一步发展和广泛应用,我们需要更精准地理解这两个基因组组装版本之间的差异和优势。
在本项研究中,毛亚飞团队整合原有算法并结合新开发的结构变异分析工具网站(SynPlotter)验证238个基因组差异区域并发现了67个新鉴定的结构差异区域
(≥10 kbp),并精确地锚定断点和结构类型。这一结果为后续的疾病诊断分子探针设计提供了精细图谱。
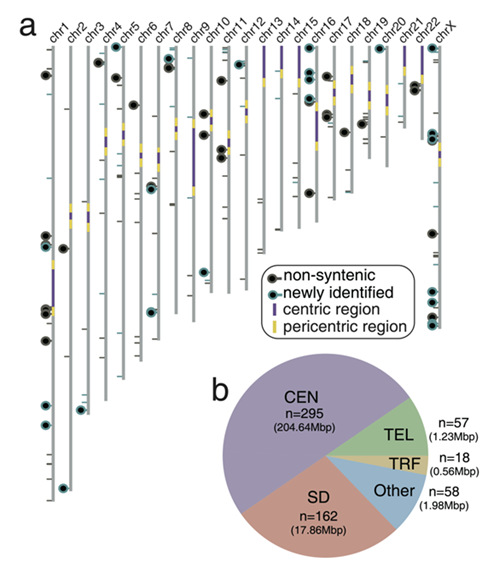
图1 GRCh38与T2T-CHM13基因组区域差异分析
该研究还发现基因组差异区域中存在着许多与生理功能相关的基因,这些基因的差异与人类脑、免疫等相关疾病息息相关。研究人员着重分析了新鉴定出的基因组差异区域中KLRC基因簇,比较了人类群体间和非人灵长类(NHP)中该基因簇的差异。以KLRC2为例,从进化、种群分型、蛋白功能及结构等多个维度,探索KLRC2的重复和缺失机制及其生理功能。
![]()
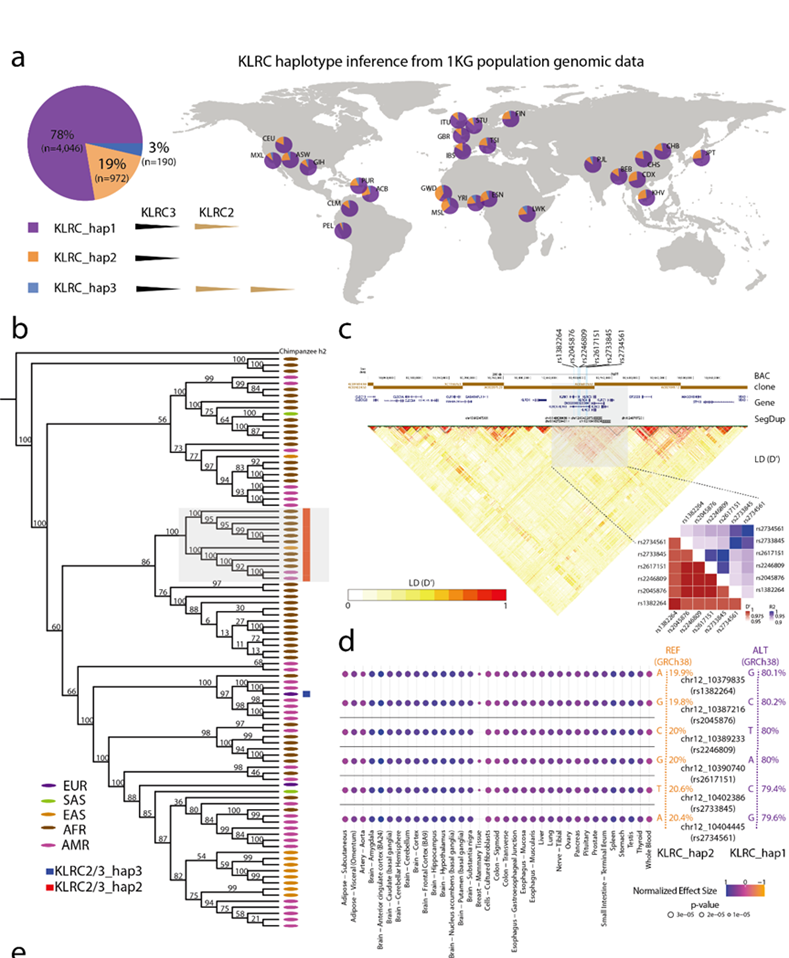
图2 人类KLRC单倍型的结构和基因功能多样性
总的来说,该项研究对人类最为重要的两个基因组模版(GRCh38和T2T-CHM13)之间大规模差异基因组区域的结构和功能进行更全面和详细评估。该结果不仅有助于我们对基因组中复杂结构遗传多样性的认识,还提出了消除参考偏差来推动未来的科学研究。研究者认为该研究的新范式在今后与HPRC (Human Pangenome Reference Consortium)、CPC (Chinese Pangenome Consortium)和Primate T2T (Telomere-to-Telomere Consortium) 产生的遗传多样性的完整基因组结合,将有助于我们充分了解人类复杂基因组片段的多样性、进化起源和功能,将极大扩展人们对复杂基因组片段的生物学认知。
本研究由上海交通大学Bio-X研究院毛亚飞实验室主导完成。上海交通大学Bio-X研究院助理研究员杨翔宇博士、硕士研究生王宣凯为本文共同第一作者。毛亚飞长聘教轨副教授为本文的通讯作者。该工作还得到了上海交通大学陆青研究员、李卫东教授,华盛顿大学Evan E. Eichler教授、约翰霍普金斯大学Michael Schatz教授等专家学者的大力支持;并得到上海交通大学“交大2030”计划,上海市浦江人才计划,国家自然科学基金青年项目等项目的资助。上海交通大学超算集群(思源一号)提供了优异的计算资源。
"交大2030"计划旨在深入贯彻习近平总书记关于科技强国建设的系列论述精神,践行高水平研究型大学使命担当。该计划致力于构建鼓励探索、宽容失败的学术文化和科研生态体系,并持续支持从事重大原创性研究的科研人员。通过改革科技创新基础制度和政策体系,建立自主支持模式,推动科研自主权的实施,并营造健康良好的科研生态环境。本课题是"交大2030"计划的首批资助项目之一,进一步凸显了该计划在推动科技创新和人才培养方面的重要性。该计划的资助使得研究团队能够在全面了解人类基因组中的大尺度结构变异方面取得突破性进展。"交大2030"计划将持续支持和推动相关重要研究,以推动科技强国建设,实现高水平科技自立自强。
原文链接:https://genomebiology.biomedcentral.com/articles/10.1186/s13059-023-02995-w
